來自美國阿貢國家實驗室、NVIDIA、芝加哥大學等組織機構的研究員開發了一個處理基因組規模數據的先進模型,并入圍戈登貝爾 COVID-19 研究特別獎決賽
這一戈登貝爾特別獎旨在表彰基于高性能計算的 COVID-19 研究。一位決賽入圍選手教會了大型語言模型(LLMs)一種新的語言——基因序列,使這些模型能夠提供基因組學、流行病學和蛋白質工程方面的洞察。
這項開創性的成果發表于 10 月,是由來自美國阿貢國家實驗室、NVIDIA、芝加哥大學等組織機構的二十多名學術和商業研究員合作完成。
該研究團隊訓練了一個 LLM 來追蹤基因突變,并預測需要關注的 SARS-CoV-2(導致 COVID-19 的病毒)變異株。雖然迄今為止大多數應用于生物學的 LLM 都是在小分子或蛋白質的數據集上訓練的,但這一項目是在原始核苷酸序列(DNA 和 RNA 的最小單位)上訓練的首批模型之一。
負責帶領該項目的阿貢國家實驗室計算生物學家 Arvind Ramanathan 表示:“我們假設從蛋白質水平到基因水平的數據有助于我們構建出更易于理解新冠病毒變異株的模型。通過訓練模型去追蹤整個基因組及其進化過程中的所有變化,我們不僅能夠更好地預測 COVID,還能預測已掌握足夠基因組數據的任何疾病。”
戈登貝爾獎被譽為 HPC 領域的諾貝爾獎。今年的戈登貝爾獎將在本周的 SC22 上由美國計算機協會頒發。該協會代表著全球約 10 萬名計算領域的專家,自2020年開始向使用 HPC 推進 COVID-19 研究的杰出研究員頒發特別獎。
在一種只有四個字母的語言上
訓練大型語言模型
長期以來,LLM 一直在接受人類語言的訓練,這些語言通常由幾十個字母組成,可以排列組合成數萬個單詞,并連接成長句和段落。而生物學語言只有四個代表核苷酸的字母,即 DNA 中的 A、T、G 和 C,或 RNA 中的 A、U、G 和 C。這些字母按不同順序排列成基因。
雖然較少的字母看似會降低 AI 學習的難度,但實際上生物學語言模型要復雜得多。這是因為人類的基因組由超過 30 億個核苷酸組成,而冠狀病毒的基因組由大約 3 萬個核苷酸組成,因此很難將基因組分解成不同、有意義的單位。
Ramanathan 表示:“在理解基因組這一‘生命代碼’的過程中,我們所面對的一個主要挑戰是基因組中的龐大測序信息。核苷酸序列的意義可能會受另一序列的影響,以人類的文本做類比,這種影響的范圍不僅僅是文本中的下一句話或下一段話,而是相當于一本書中的整個章節。”
參與該項目協作的 NVIDIA 研究員設計了一種分層擴散方法,使 LLM 能夠將約 1500 個核苷酸的長字符串當作句子來處理。
論文共同作者、NVIDIA AI 研究高級總監、加州理工學院計算+數學科學系布倫講席教授 Anima Anandkumar 表示:“標準語言模型難以生成連貫的長序列,也難以學習不同變異株的基本分布。我們開發了一個在更高細節水平上運作的擴散模型,該模型使我們能夠生成現實中的變異株,并采集到更完善的統計數據。”
預測需要關注的新冠病毒變異株
該團隊首先使用細菌和病毒生物信息學資源中心的開源數據,對來自原核生物(像細菌一樣的單細胞生物)超過 1.1 億個基因序列進行了 LLM 預訓練,然后使用 150 萬個高質量的新冠病毒基因組序列,對該模型進行微調。
研究員還通過在更廣泛的數據集上進行預訓練,確保其模型能夠在未來的項目中推廣到其他預測任務,使其成為首批具備此能力的全基因組規模的模型之一。
在對 COVID 數據進行了微調后,LLM 就能夠區分病毒變異株的基因組序列。它還能夠生成自己的核苷酸序列,預測 COVID 基因組的潛在突變,這可以幫助科學家預測未來需要關注的變異株。
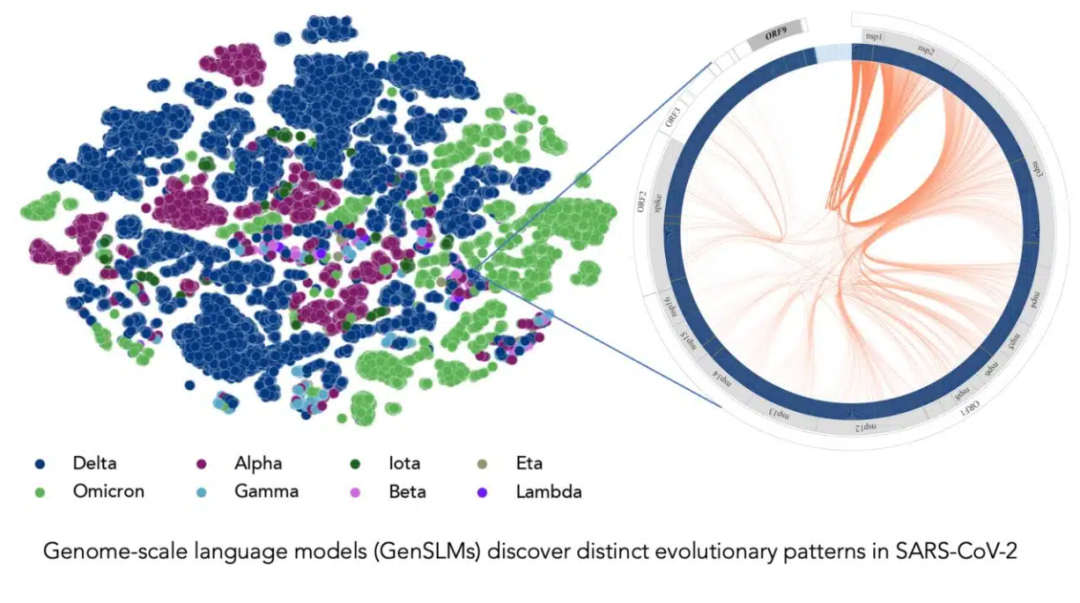
在長達一年時間內積累的 SARS-CoV-2 基因組數據的訓練下,該模型可以推斷出各種病毒株之間的區別。左邊的每個點對應一個已測序的 SARS-CoV-2 病毒株,并按變異株顏色編碼。右圖放大了該病毒的一個特定毒株,它捕捉到了該毒株特有的病毒蛋白進化耦合關系。圖片由美國阿貢國家實驗室的 Bharat Kale、Max Zvyagin 和 Michael E. Papka 提供。
Ramanathan 表示:“大多數研究員一直在追蹤新冠病毒突刺蛋白的突變,尤其是與人類細胞結合的域。但病毒基因組中還有其他蛋白質也會經歷頻繁的突變,所以了解這些蛋白質十分重要。”
論文中提到,該模型還可以與 AlphaFold、OpenFold 等常見的蛋白質結構預測模型整合,幫助研究員模擬病毒結構,研究基因突變如何影響病毒感染其宿主的能力。OpenFold 是 NVIDIA BioNeMo LLM 服務中包含的預訓練語言模型之一。NVIDIA BioNeMo LLM 服務面向的是致力于將 LLM 應用于數字生物學和化學應用的開發者。
利用 GPU 加速超級計算機
大幅加快 AI 訓練速度
該團隊在由 NVIDIA A100 Tensor Core GPU 驅動的超級計算機上開發 AI 模型,包括阿貢國家實驗室的 Polaris、美國能源部的 Perlmutter 以及 NVIDIA 的 Selene 系統。通過擴展到這些強大的系統,他們在訓練中實現了超過 1500 exaflops 的性能,創建了迄今為止最大的生物語言模型。
Ramanathan 表示:“我們如今處理的模型有多達 250 億個參數,預計這一數量未來還會大幅增加。模型的尺寸、基因序列的長度、以及所需的訓練數據量,都意味著我們的確需要搭載數千顆 GPU 的超級計算機來完成復雜的計算。”
研究員估計,訓練一個具有 25 億參數的模型版本,需要約 4000 個 GPU 耗時一個多月。該團隊已經在研究用于生物學的 LLM,在公布論文和代碼之前,他們在這個項目上已耗時約四個月。GitHub 頁面上有供其他研究員在 Polaris 和 Perlmutter 上運行該模型的說明。
NVIDIA BioNeMo 框架可在 NVIDIA NGC 中心上的 GPU 優化軟件中搶先體驗。該框架將幫助研究員在多個 GPU 上擴展大型生物分子語言模型。作為 NVIDIA Clara Discovery 藥物研發工具集的一部分,該框架將支持化學、蛋白質、DNA 和 RNA 數據格式。
即刻點擊“閱讀原文”或掃描下方海報二維碼,收下這份 GTC22 精選演講合集清單,在NVIDIA on-Demand 上點播觀看主題演講精選、中國精選、元宇宙應用領域與全球各行業及領域的最新成果!
原文標題:SC22 | 解析基因組的“語言”:戈登貝爾獎決賽選手使用大型語言模型來預測新冠病毒變異株
文章出處:【微信公眾號:NVIDIA英偉達企業解決方案】歡迎添加關注!文章轉載請注明出處。
-
英偉達
+關注
關注
22文章
3950瀏覽量
93725
原文標題:SC22 | 解析基因組的“語言”:戈登貝爾獎決賽選手使用大型語言模型來預測新冠病毒變異株
文章出處:【微信號:NVIDIA-Enterprise,微信公眾號:NVIDIA英偉達企業解決方案】歡迎添加關注!文章轉載請注明出處。
發布評論請先 登錄
中科曙光構建全國產化基因組學高性能計算平臺
?VLM(視覺語言模型)?詳細解析
無法在OVMS上運行來自Meta的大型語言模型 (LLM),為什么?
小白學大模型:訓練大語言模型的深度指南

大語言模型的解碼策略與關鍵優化總結

大語言模型開發語言是什么
云端語言模型開發方法
大語言模型如何開發
如何利用大型語言模型驅動的搜索為公司創造價值

【《大語言模型應用指南》閱讀體驗】+ 基礎篇
DeepL推出新一代翻譯編輯大型語言模型
基于CPU的大型語言模型推理實驗






 SC22 | 解析基因組的“語言”:戈登貝爾獎決賽選手使用大型語言模型來預測新冠病毒變異株
SC22 | 解析基因組的“語言”:戈登貝爾獎決賽選手使用大型語言模型來預測新冠病毒變異株











評論